- 寨卡病毒病由通过伊蚊传播的一种病毒引起。
- 寨卡病毒病患者的症状可能包括轻微发热、皮疹、结膜炎、肌肉和关节疼痛、浑身虚弱或头痛。
- 这些症状通常持续2-7天。科学界的共识是寨卡病毒是小头症和吉兰–巴雷综合征的一个病因。
尽管目前对于寨卡病毒的理解逐渐加深,但目前还没有可用的抗病毒疗法。最近报道的化合物能够阻止寨卡病毒在神经细胞中的恶化,但没有报道其溶解性,药物靶点机制,以及体内反应。因此,急需理解其传染机制,进而通过多重策略来狙击寨卡病毒的传播。
寨卡病毒的基因组位于衣壳内部,含有一条10794kb的单链正义RNA序列,包括2个非编码区(5’和3’非编码区)以及一个编码多聚蛋白的开放式阅读框。多聚蛋白由7个非结构蛋白(NS)组成,其在RNA复制中扮演着重要角色。非结构蛋白5是(NS5,~103 kDa)最大的,最保守的病毒蛋白。NS5由一个有甲基转移酶活性(MTase)的N端RNA帽子结构域和一个RNA依赖性的C端RNA聚合酶(RdRp)结构域组成。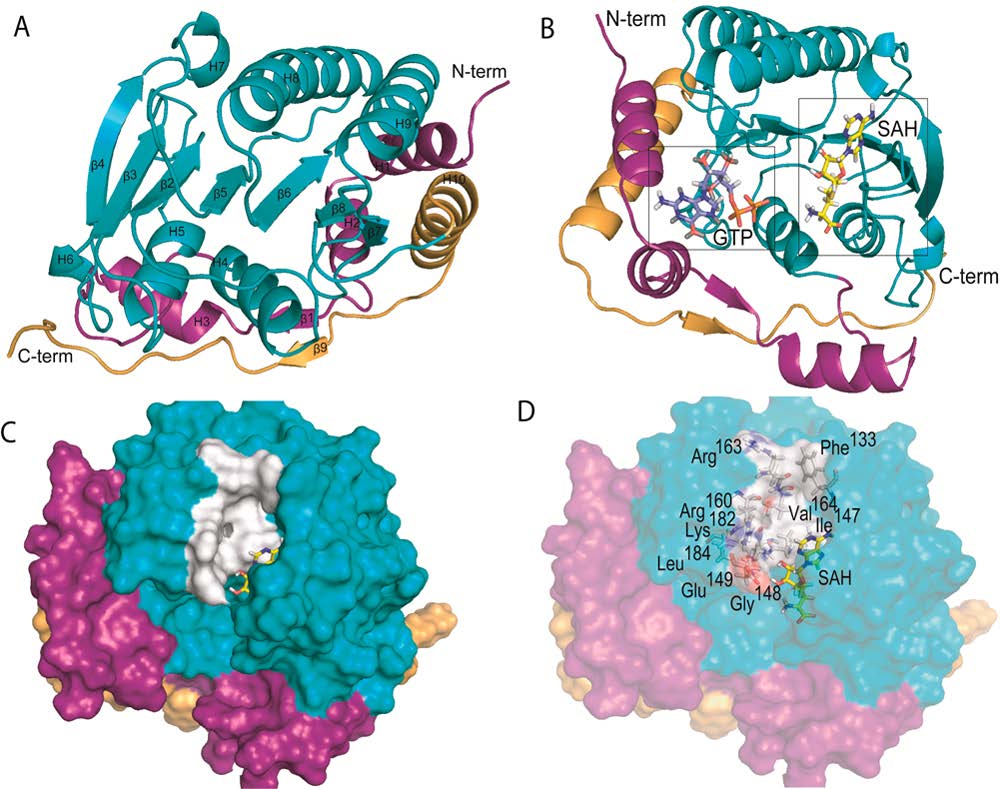
目前,在黄病毒中,基于NS5的药物设计来针对MTase和RdRp已经开始尝试。针对ZIKV RdRp新型核苷酸类似物已经被报道。RdRp金属结合口袋和MTase的SAH/SAM(S-adenosyl-homocysteine/S-adenosyl-L-methionine)结合口袋已经用于药物筛选。另一个提出的靶点在NS5,如针对病毒复制过程中的NS3和NS5相互作用。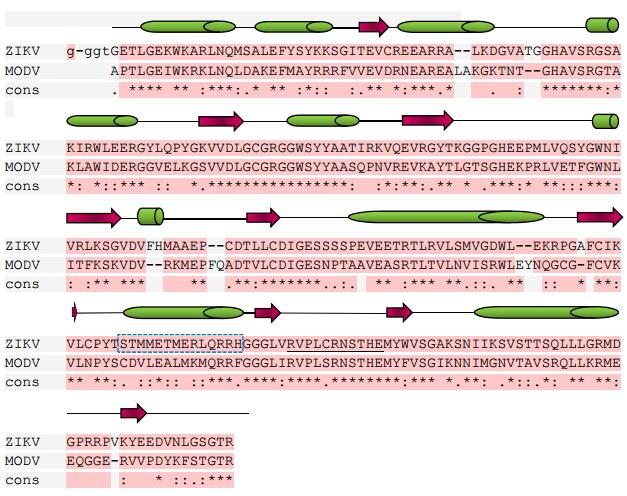
黄病毒MTase有一个临近SAH/SAM结合口袋的保守的疏水腔。而人体N7/2′OMTase中,缺乏这些保守的疏水性结合口袋。目前,ZIKV的MTase晶体结构已经被解析出来,但这个项目开始时还没解析出来。因此,作者以Modoc病毒的MTase结构(MODV; PDB ID:2WA1)作为模板,构建了MTase三维结构(Modeler 9.14). MODC的MTase结构是PDB数据库中唯一一个没有配体结合晶体结构。SwissPDB viewer中的最速下降法被用于能量最小化。该模建结构存储在PMDB数据库中,ID为PM0080437。模建的ZIKV NS5模型和已解析出来的晶体结构是一致的,都包含43个阿尔法螺旋和20个贝塔片层结构。
辅因子SAM在黄病毒中是甲基供体,它转移甲基到酶底物中,并被转化成SAH。作者用SwissDock获得了ZIKV MTase SAH复合物,而GOLD的遗传算法获得了ZIKV MTase GTP复合物。
虚拟筛选
software: GOLD 5.4.1
database:28 341 compounds from Life Chemicals database
binding pocker:Ten Ångstroms around Val164
(This was to derive the best possible conformers utilizing the hydrophobic region near to SAM binding pocket.)
criteria:the Lipinsky’s rule of five, Veber criteria, and dissimilarity evaluation.
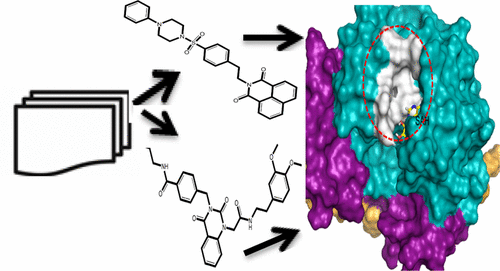
The best 10 ligands were chosen based on the scores obtained from the ChemPLP and verifying with the genetic docking algorithm GOLD.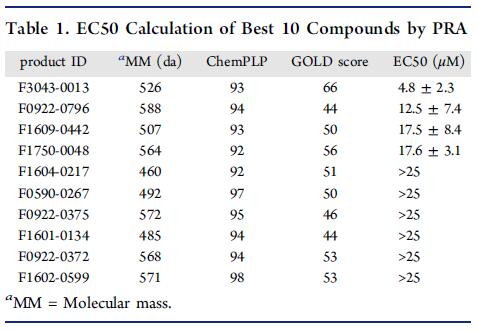
GOLD score is based on H-bonding energy, van der Waals energy,
metal interaction, and ligand torsion strain, whereas ChemPLP
uses the hydrogen bonding term and multiple linear potentials to
model van der Waals and repulsive terms.
Plaque reduction assay (PRA), a gold standard phenotypic method for in vitro efficacy analysis was carried out by treating each of the 10 compounds against ZIKV isolated from a Canadian traveler in 2013.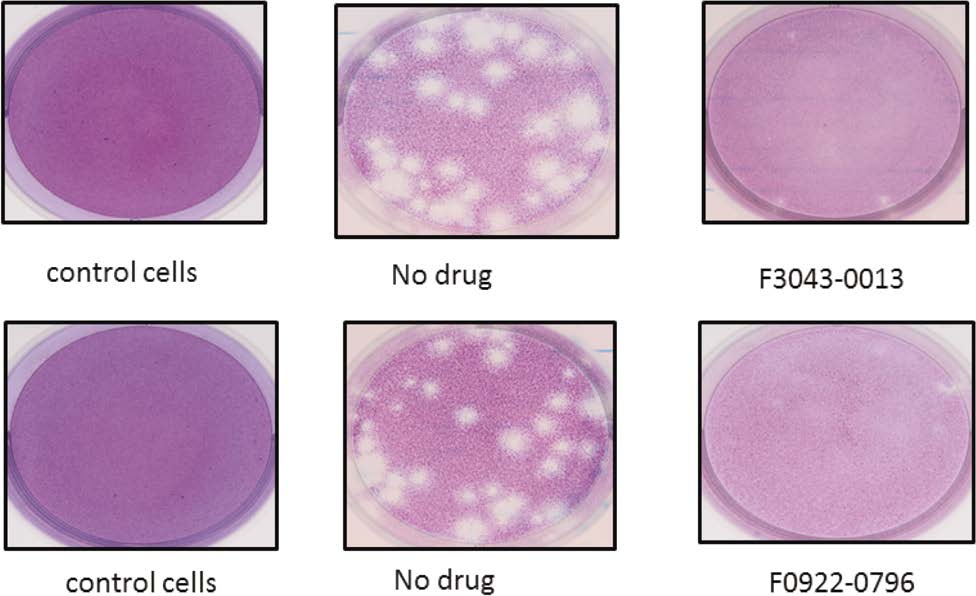
蛋白-配体结合模式图。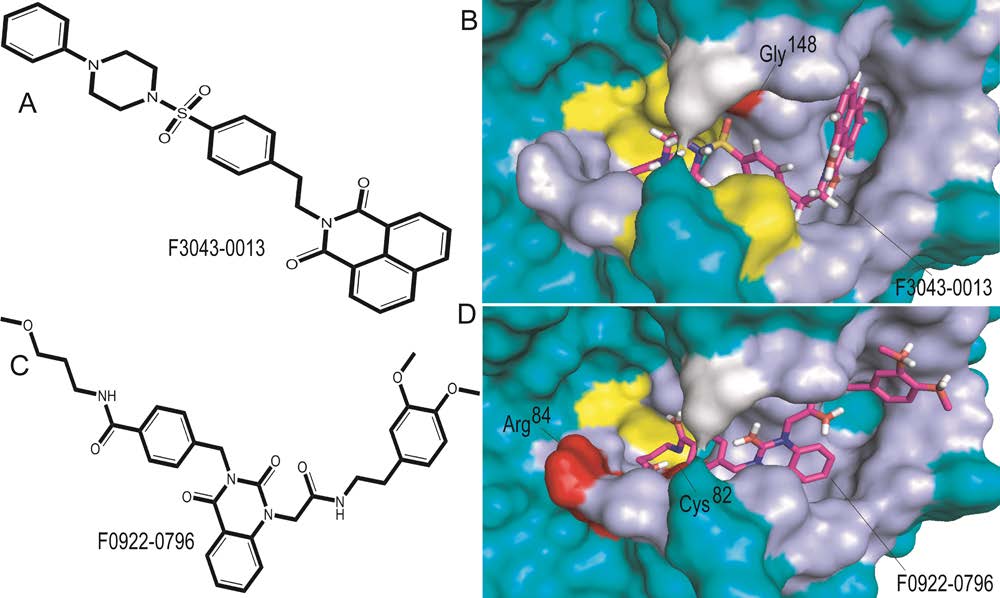
实验方法:
1.Sequence/structure Alignment
2.Model Building and refinement
3.Substrate docking
4.Virtual screening
5.Mutation and structure analysis
6.Plaque reduction assay (PRA)
Reference
Structural Insight into NS5 of Zika Virus Leading to the Discovery of MTase Inhibitors.链接:http://pubs.acs.org/doi/abs/10.1021/jacs.6b10399