我们在做动力学模拟的时候,经常发现蛋白质晶体结构中缺失loop,这是因为在蛋白质晶体结构解析的过程中,柔性较高的loop无法准确测定所导致的。下面我们将使用chimera+modeller来补缺丢失的残基。
软件:chimera,modeller最新版
材料:缺失loop区的蛋白质晶体结构,蛋白质完整序列(fasta格式)
方法:1.打开chimera,载入pdb文件,可以看到loop区结构缺失,红色标记部分。见下图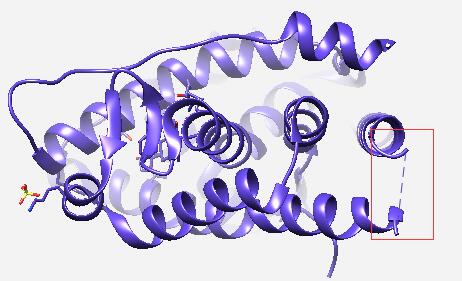
2.载入fasta序列,如下图所示。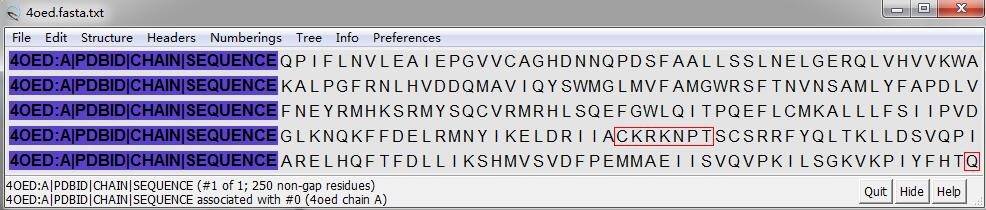
3.点击序列界面中的structure,选择modeller(loop/refinement),按照下图所示的方法设置,然后点击apply。从设置好的生成路径中选择B99990001.pdb即可。
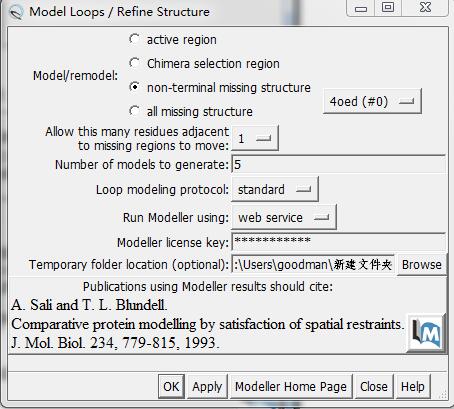
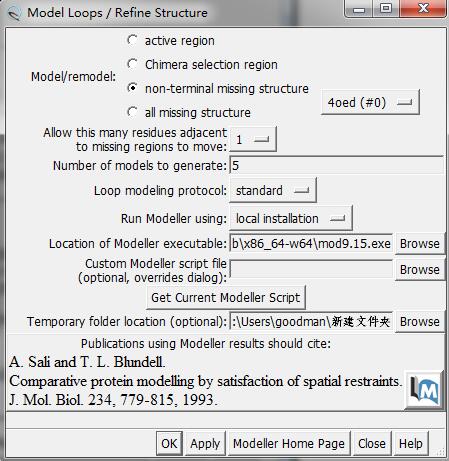
注:以上使用了web和local两种方法来补全残基,发现有差别,但整体差别可以忽略不计。其中,local可以在本地文件中直接找到,而web中的本地文件只是残缺部分。故推荐使用local法。除使用本方法外,还可以使用Schrodinger中的蛋白准备来补缺残基
本文参考借鉴了BioMS阿里原创教程“巧用chimera1.7进行晶体结构缺失部分的修补”。